Vergleicht man nicht die Aminosäuresequenzen, sondern direkt die Nukleotidsequenzen verschiedener Arten miteinander, so erhält man für den Genabschnitt des Cytochrom c die folgende Anzahl unterschiedlicher Nukleotide bei verschiedenen Arten:
| Verglichene Arten: |
Anzahl der unterschiedlichen Nukleotide: |
| Mensch-Schwein | 13 |
| Mensch-Ente | 17 |
| Mensch-Klapperschlange | 20 |
| Mensch-Thunfisch | 31 |
| Mensch-Motte | 36 |
| Mensch-Hefe | 66 |
⇒ Je höher die Anzahl der unterschiedlichen Nukleotide, desto
weiter liegt die Verzweigung im Stammbaum zurück
und desto weitläufiger ist die Verwandtschaft.
und desto weitläufiger ist die Verwandtschaft.
Hieraus ergibt sich der folgende Stammbaum:
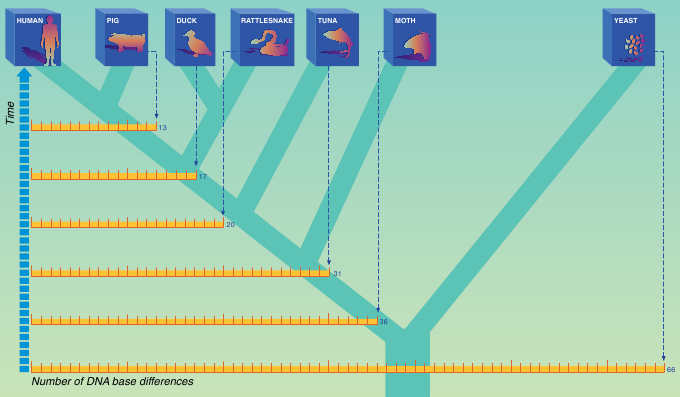
Beachte, dass die Anzahl der substituierten Nukleotide größer ist (z.B. Mensch-Hefe: 66) als die Anzahl der veränderten Aminosäuren (z.B. Mensch-Bäckerhefe: 44)!
⇒ Aufgrund der Degeneration des genetischen Codes sind Nukleotidsequenzvergleiche
zuverlässiger als Aminosäuresequenzvergleiche.
Weitere Informationen zum Zusammenhang zwischen der Nukleotidsequenz und der Aminosäuresequenz aufgrund
des genetischen Codes sowie dessen Degeneration
findet man in den beiden Lernprogrammen
Translator und
Reverse Translator.
Zum Weiterlesen:
- Systematics and Molecular Phylogenetics
- PhylogeneticTrees
- Interactive Tree of Life
- Bioinformatics with pen and paper: building a phylogenetic tree